다양한 의료영상 가운데 많이 사용되고 있는 있는 MRI는 촬영 방식에 따라 종류가 다양합니다. 이 논문은 촬영된 MRI들을 가지고 다른 contrast를 가지는 MRI를 생성하는 논문입니다.
새로운 contrast의 MRI를 생성하도록 특화된 네트워크 (CC-DNN)과 더불어, misregistration를 보정하고 noise에 덜민감하도록 만드는 loss를 추가함으로써 뛰어난 성능을 보여줍니다.
해당 논문은 MRI분야 논문지인 Magnetic Resonance in Medicine에 게재되었습니다.
https://onlinelibrary.wiley.com/doi/10.1002/mrm.28327
Introduction
MRI는 다양한 pulse sequence를 통해 필요한 부위를 특화시켜 볼 수 있는 가장 유명한 진단 툴입니다.
다양한 종류의 MRI가 존재하는데 그 가운데 가장 많이 촬영하는 영상은 spin-ehco 방식의 T1-weighted (T1w), T2-weighted (T2w), PD-weighted 영상과, gradient-echo 방식의 GRE 영상이 있습니다.
그리고 이 논문에서 다룰 부위인 무릎쪽에서는 fat suppression image인 short tau inversion recovery (STIR)이 가장 많이 촬영됩니다.

위와 같이 같은 영역이라도 어떤 sequence로 촬영하느냐에 따라 다른 contrast의 영상이 생성됩니다.
물론 여러장의 다양한 contrast의 MRI 영상이 있다면 진단의 효율을 상승시킬 수 있지만, 모든 영상을 동시에 촬영하는 것이 아니고 순차적으로 촬영을 해야하기 때문에 시간이 오래 소요됩니다.
한장의 MRI를 획득하는 시간은 보통 3~6분 정도이기 때문에 여러장을 촬영하다보면 병원의 환자 회전율도 떨어지고 환자도 오래동안 MRI 기계안에 있어야하기 때문에 움직임이 들어갈 확률이 높아져 영상의 quality가 떨어질 수 있습니다.
그러므로 촬영된 몇장의 MRI를 통해 새로운 contrast 영상을 만들어내는 synthetic MRI 분야가 요구되고 연구도 활발히 진행되고 있습니다.
이 논문 역시 T1w, T2w, GRE 영상을 가지고 contrast conversion에 특화된 뉴럴네트워크 구조와, 새로운 loss function을 통해 STIR 영상을 만들어내는 연구입니다.
Dataset
논문에서는 연구의 가능성을 살펴보기 위해 실제 MRI 데이터를 가지고 실험하기 전 simulation data를 만든 후 실험을 진행했습니다.
1. MR Physics

위의 식들은 실제 MRI를 생성하는 식으로 bloch equation이라고 부릅니다.
먼저, TR과 TE는 촬영하고자 하는 영상이 무엇인지에 따라 결정되는 파라미터로 MRI 기계에 촬영시 입력하는 값입니다.
반면, S0, T1, T2, T2*은 신체조직이 가지고 있는 고유값에 해당합니다. 예를들어 물의 T1 =4000, T2=2000이고 근육의 T1=900, T2=50입니다. 이와같이 각 조직별로 고유의 값이 있기때문에 TR, TE를 조절하면서 원하는 부분의 contrast를 밝게하고, 보고싶지 않은 부분은 어둡게 만들수 있습니다.
즉, TR, TE를 조절하면서 특정부위를 강조할 수 있도록 위와 같은 다양한 식들이 만들어졌고, 해당 식을 통해 다양한 영상들이 생성되고 있습니다.
2. Simulation Data
MRI를 구성하는 식을 알고 있고, 실제 데이터셋을 촬영할때 사용한 TR, TE 값을 알고 있기 때문에, simulation 데이터를 직접 생성할 수 있습니다.
신체 조직이 가질 수 있는 조합을 고려하여 무수히 많은 데이터를 생성했습니다.
S0 : 0.01 ~ 1 (0.01 간격)
T1 : 10 ~ 4000 (10 간격)
T2 : 10 ~ 2000 (10 간격)
T2* : 10 ~ 1000 (10 간격)
* T2* < T2 < T1

위 그림처럼 T1, T2, T2*, S0 맵을 만든 뒤, bloch equation을 통해 T1w, T2w, GRE, STIR 영상을 생성했습니다.
3. In-vivo Data
총 12명의 건강한 실험자를 모집하여, 3T Philips MRI scanner로 촬영하여 데이터를 획득했습니다.
T1w : TR = 522, TE = 11, 촬영시간 =2분 39초
T2w : TR = 3000, TE = 80, 촬영시간 = 3분 12초
GRE : TR = 500, TE = 60, flip angle(θ) = 25°, 촬영시간 = 3분 44초
STIR : TR = 4478, TE = 60, TI = 220, 촬영시간 = 4분 38초
384x384 resolution으로 한명당 24 slice를 촬영하여 전체 288장의 데이터셋을 구축하였습니다. Train / Test로 12명을 9/3로 나눴고, 4-fold cross validation을 통해 모든 데이터를 활용했습니다.
Network Architecture

위 그림은 논문에서 고안한 Contrast Conversion-Deep Neural Network (CC-DNN) 입니다.
먼저 세 장의 input MRI 영상이 들어와 병렬적으로 convolution layer를 거쳐 학습을 진행합니다. 처음부터 합쳐서 학습을 진행하는 것이 아니고 병렬적으로 진행하면서, 각 영상별로 중요한 feature들을 먼저 추출하도록 유도하였습니다.
그리고 병렬적으로 추출된 feature들은 concatenate layer를 통해 하나로 합쳐준 뒤, 다시 convolution layer를 거처 label인 STIR 영상을 생성하도록 학습합니다.
Residual block과 skip connection을 활용해 네트워크를 구성하였습니다.
Loss Function
논문에서는 총 세개의 loss를 제안했습니다.
1. MSE loss

영상을 재구성하는데 가장많이 사용하는 loss로서 label과 output 픽셀간 차이의 제곱으로 계산됩니다.
2. Misregistration loss
in-vivo 데이터셋은 실험자가 MRI 기계 안에서 총 4가지의 영상을 촬영하여 획득한 것입니다. 실험전 실험자에게 촬영시간 동안 움직임을 최소화해달라고 당부하였고, 실제 MRI 스캐너도 움직임을 보정하여 영상을 재구성합니다.
하지만 완벽하게 4장의 MRI가 동일한 픽셀위치에 생성되는것을 불가능합니다.
MSE loss는 픽셀간의 값 차이를 통해 계산되기 때문에, 위치 차이가 존재한다면 값을 잘못 계산하거나 blurring을 만드는 등 학습에 악영향을 끼칠 수 있습니다.

그러므로 misregistration을 보완하기 위해 위와 같은 loss를 추가하였습니다.
해당 loss는 동일한 위치의 pixel값의 차이를 바로 loss로 사용하는 것이 아닌, k x k 주위의 픽셀가운데 가장 값차이가 적은 부분의 값을 loss로 사용하는 것입니다. 즉, 미세하게 픽셀의 위치가 달라졌을 경우라도 해당 위치를 찾아서 loss를 계산하기 때문에 학습 오류를 최소화 할수 있습니다.
3. Variation loss

이 loss 역시 MSE의 단점을 보완해주기 위해 고안된 loss로서, 픽셀간 차이만 고려하는 것이 아닌 주변 픽셀간의 분산(variation)까지 고려하기 위한 loss입니다. noise가 추가된 경우 픽셀값만 가지고 계산하다보면 오차가 생길 위험이 커지기 때문에, 주변값들의 분산을 활용해 noise가 학습에 악영향을 끼치는것을 최소화 할 수 있습니다.
Results
1. Simulation Data Result

위 그림은 simulation dataset을 가지고 CC-DNN을 학습한 결과입니다. 실제 MRI 촬영도중 추가되는 Rician noise를 추가했음에도 STIR의 contrast를 잘 생성하는 것을 확인할 수 있습니다.
2. Added Loss Effect

위 그림은 misregistration loss와 variation loss의 성능을 보여줍니다.
먼저 Case1은 input으로 들어가는 T1w, T2w, GRE을 상하좌우로 랜덤하게 1~2 pixel씩 흔든 이미지를 가지고 학습을 진행한 결과입니다. MSE loss (c1)만 사용한 결과영상(A)를 보면 경계가 뭉그러지는 것을 확인 할 수 있습니다.
반면에, misregistration loss를 함께 사용한 결과영상(B)를 보면 경계가 분명해짐을 확인 할 수 있습니다.
Case2는 input으로 들어가는 T1w, T2w, GRE에 noise를 섞은 이미지를 가지고 학습을 진행한 결과입니다. MSE loss (c1)만 사용한 결과영상(D)를 보면 contrast차이가 크지 않은 경계부분에 noise까지 추가되다보니 경계가 불분명해지고 뭉그러지는 것을 확인 할 수 있습니다. 반면에, variation loss를 함께 사용한 결과영상(E)를 보면 분산까지 고려하기 때문에 noise영향을 덜받게되어 경계가 분명해짐을 확인 할 수 있습니다.
3. In-vivo Data Result
in-vivo data는 MRI 촬영시 발생하는 주변요소들과, 기계안에서 처리하는 보정작업 등 단순한 bloch equation만으로 생성되는 영상이 아닙니다. 그러므로 bloch equation 기반 simulation data에서 결과가 잘 나올지라도 in-vivo data를 통해 검증을 반드시 해야만 합니다.

위 그림은 in-vivo data에 대한 CC-DNN의 결과입니다. STIR 영상에서 가장 중점적으로 살펴보는 부분인 cartilage, meniscus, tendon, acl에서 실제 획득한 STIR(빨강)와 CC-DNN으로 생성된 STIR(파랑)의 contrast가 거의 정확함을 확인할 수 있습니다. 이는 네트워크가 bloch equation 뿐만 아니라 외부요인까지 고려하도록 학습이된것을 확인할 수 있습니다.

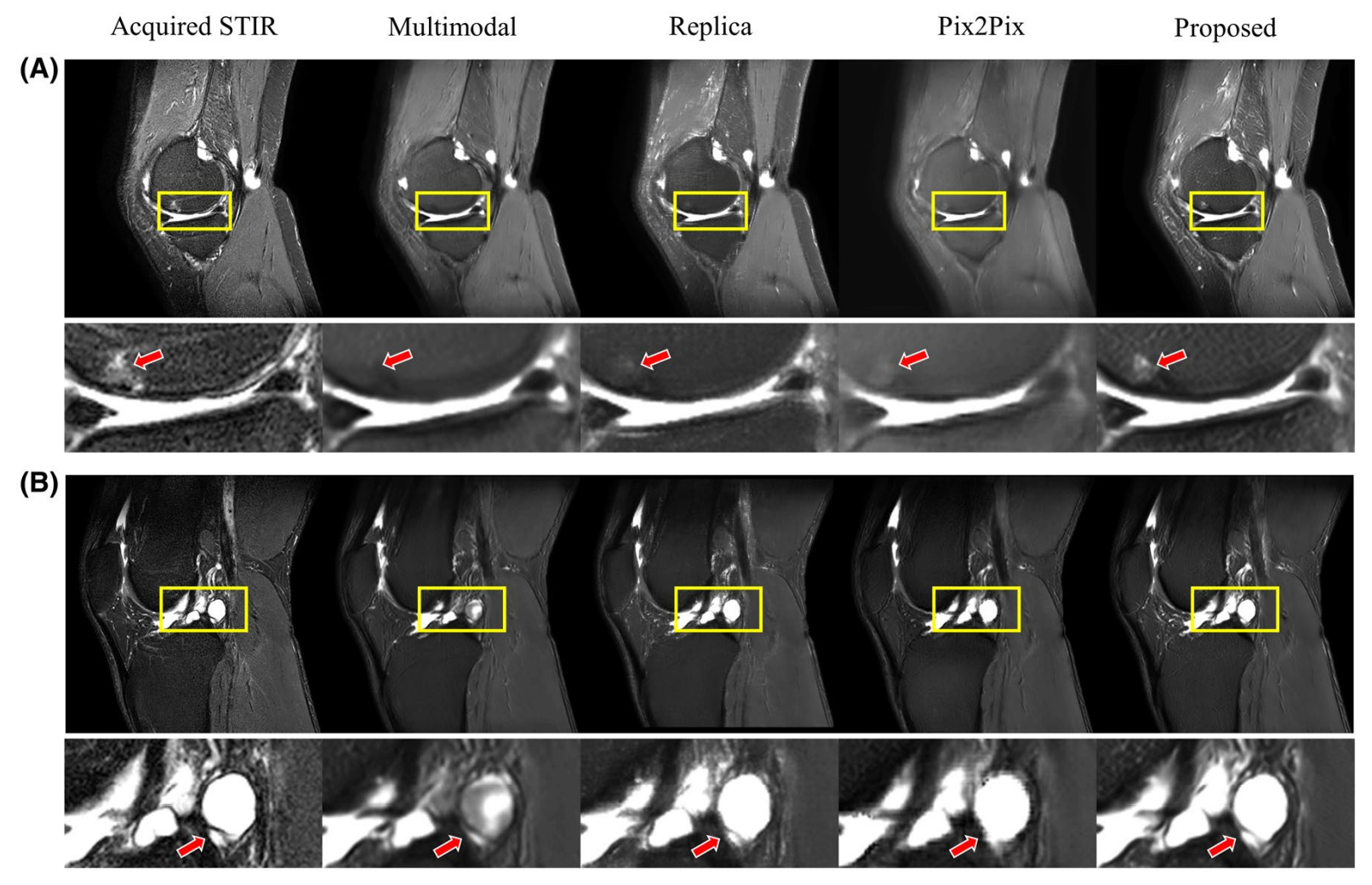
위 그림은 기존의 synthetic MRI의 기술들과의 비교 결과입니다. 실제 영상의학과 의사들이 집중적으로 보는 detail한 영역에 대해 다른 기술들과 비교하여 CC-DNN이 좋은 결과를 보임을 확인 할 수 있습니다.

영상뿐만 아니라 객관적 평가 metric에서도 다른 기술들과 비교하여 수치상에서 좋은 결과를 보임을 확인 할 수 있습니다.
마지막으로, 학습에 사용되지 않은 환자 데이터로 테스트한 결과입니다.
학습시에는 건강한 실험자들의 영상들로만 학습을 진행했기에, 네트워크가 환자 영상을 학습하지는 않았습니다.
그러나, 실질적으로 CC-DNN은 T1w, T2w, GRE의 pixel값 조합이 STIR의 pixel 값으로 변하는 함수를 학습하게 됩니다. 그러므로 실제 본적없는 edema(부종)같은 것들도 얼추 비슷하게 생성하는 것을 확인 할 수 있습니다.
이를 통해 환자 데이터를 학습에 추가한다면, 성능면에서는 더 뛰어난 결과를 보일 것으로 예상됩니다.
댓글